Unik markörresurs effektiviserar framtidens tallförädling

Genomisk selektion är en ny förädlingsmetod som utnyttjar DNA-markörer istället för stamtavlor vid utvärdering av kandidatträd. De bästa kandidaterna väljs sedan som föräldrar till nästa generationens förädlingsträd och som föräldraträd i storskaliga fröodlingar. Med DNA-markörer går det potentiellt att göra nogrannare genetiska prediktioner än med stamtavlor. Det beror på att stamtavlorna inte speglar det faktiska släktskapet mellan individer och kan vara missvisande, bland annat på grund av pollenkontamination eller felaktig identifikation.
För att göra genomisk selektion effektivt krävs en bra markörresurs som är baserad på sekvensering av hela genomet. Forskare på Umeå Plant Science Center och Uppsala universitet har nu sekvenserat tallgenomet och producerat ett så kallat SNP-chip, som är en unik markörresurs för inhemsk tall. Forskningsresultatet visade att man även kan använda denna markörresurs för andra träd i Pinus-släktet, till exempel contortatall.
Bakgrund
Förädlingen av tall påbörjades i Sverige på 1940-talet med att särskilt fina träd från skogsbestånd valdes till att bli nästa generationens föräldrar. Därefter har förädlingsarbetet uppvisat ökade genetiska vinster för varje ny mätning och nytt urval som genomförts. Traditionellt har genetiska avelsvärden för varje träd beräknats med en hjälp av en kombination av mätdata av intressanta egenskaper hos träden (till exempel höjdtillväxt) och trädens inbördes släktskap genom deras stamtavla (klassisk avelsvärdering). Men det är också möjligt att läsa av väl valda delar av trädindividernas DNA med genomiska markörer. Informationen från dessa kan således ge en lika bra eller ännu bättre bild av det faktiska släktskapet mellan individer än stamtavlan, förutsatt att man använder sig av tillräckligt många och bra markörer. Genomisk kartläggning med hjälp av markörer (populärt kallat genotypning) kan således ersätta stamtavlan vid avelsvärderingen. Avelsvärden från en sådan analys kan till och med ha större träffsäkerhet än traditionella avelsvärden, särskilt om markörresurserna är tätt kopplade till genetiken bakom de egenskaper som önskas.
Så kallade SNPs (Single Nucleotide Polymorphism) är för närvarande de mest populära typerna av markörer för att genotypa individer inom förädlingsforskningen. En sådan markör läser av variationen i en DNA-bas på ett enda specifikt ställe i genomet. Informationen från varje enskild SNP är således mycket begränsad, men genom att läsa en mycket stor mängd sådana markörer på många olika ställen i genomet får man en mycket bättre bild av släktskap och den genetiska arkitekturen hos varje kartlagd individ. För att möjliggöra storskalig genotypning (många SNPs och många individer), organiseras genotypningen så att en stor uppsättning markörer samplas i en enda bioteknisk avläsningspanel, ett så kallat SNP-chip. Dessa SNP-chip måste designas på förhand för den art (ibland flera arter) man vill genotypa storskaligt.
SNP-chip för tall utvecklades
I denna studie utvecklades ett SNP-chip för tall genom att sekvensera 192 träd. Av dessa träd, hade 172 sitt ursprung från olika förädlingspopulationer runt om i landet, medan 20 träd representerade naturligt proveniensmaterial i Sverige. Därefter genotypades 480 träd, vilka hade ursprung över hela tallens utbredningsområde i Eurasien, med avseende på ett mycket stort antal SNP-markörer (430 000 SNPs samlade i ett mellansteg, ett så kallat ”utvecklingschip”). De flesta av dessa träd (301 st) hade sitt ursprung i förädlingspopulationer från mellersta och norra Sverige, men resten kom från proveniensförsök i Tjeckien (74), och ytterligare träd från naturliga populationer från Tjeckien (3), Finland (2), Skottland (2) och Spanien (3). Ytterligare sex familjekombinationer av modern, fadern och sju till åtta avkommor per familj genotypades så att SNP-markörernas kvalitet kunde valideras och säkras.
DNA-prover samlades även från andra utländska tallarter för att utreda vilka SNP-markörer som var användbara även för dessa arter. Således provtogs totalt 192 träd från arter såsom Banksianatall (Pinus banksiana Lamb.), cembratall (P. cembra L.), contortatall (P. contorta, (Dougl.)), svarttall (P. nigra Arnold), och weymouthtall (P. strobus L.) samt sydkinesisk tall (P. tabuliformis (Carr)). De sydkinesiska tallproverna insamlades i Kina av Beijing Forestry University medan alla andra arter samlades från Arboretum Sofronka i Tjeckien.
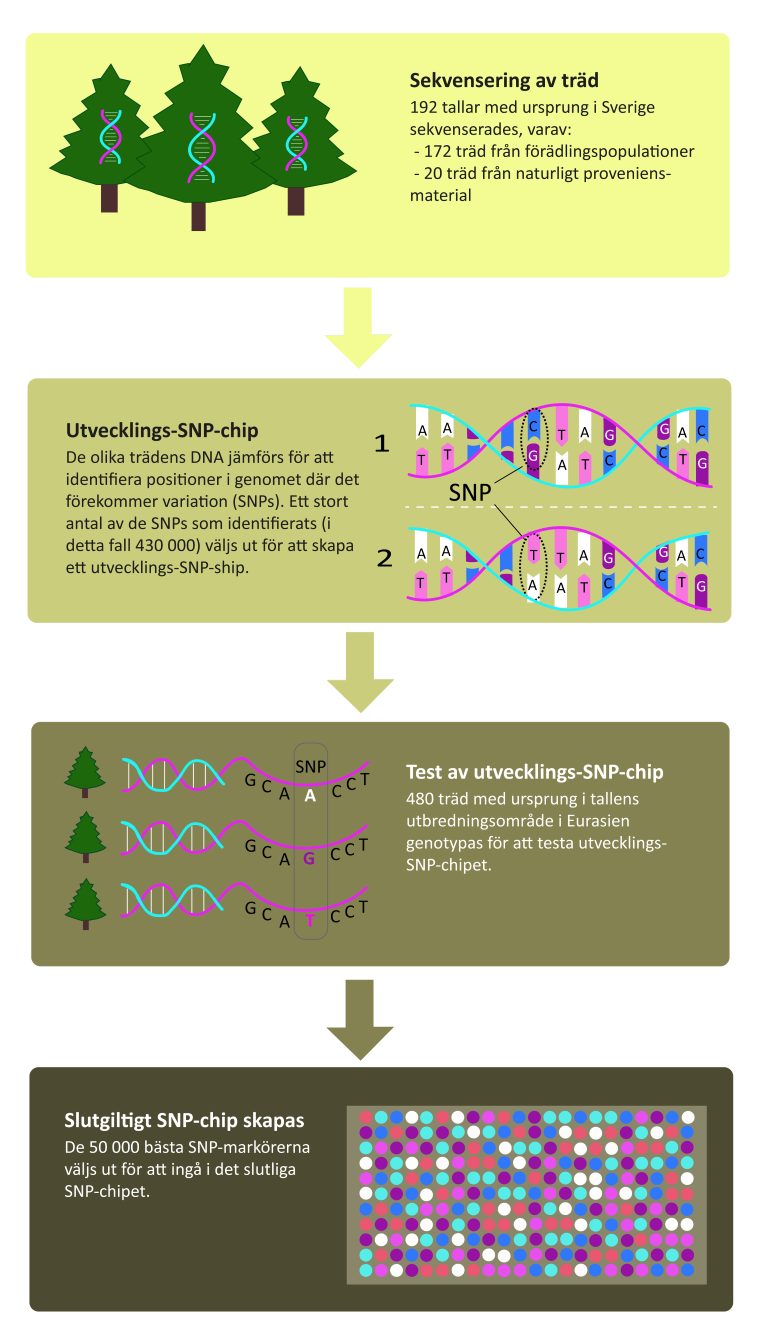 Figur 1. Översiktlig bild av utvecklingsprocessen för SNP-chip för tall.
Figur 1. Översiktlig bild av utvecklingsprocessen för SNP-chip för tall.
Urval i flera steg
Urvalet av de 50 000 markörer som skulle ingå i det slutliga SNP-chipet, innefattade flera steg. Validering och utvärdering baserades på kvalitetsmått såsom markörernas informationsmässighet, pålitlighet, fördelning över tallgenomet, samt associationer med miljön och med egenskaper viktiga i förädlingsurvalet. Endast de mest informativa och relevanta SNP-markörerna valdes ut att ingå i det slutliga chipet. Markörer som låg väldigt nära varandra i tallgenomet kunde vara nära länkade och korrelerade med varandra, och i dessa fall valde man enbart ut en av dem till chipet. Flera SNP-markörer som var associerade med egenskaper av intresse i förädling, som till exempel diameter, höjd, kvistantal, överlevnad och stamvolym, valdes att ingå SNP-chipet. Vissa markörer var även associerade med trädens geografiska ursprung såsom ursprungslatitud, -longitud och -altitud.
Släktskapsförhållanden mellan individer fångades tydligt med hjälp av de utvalda markörerna. Var och en av de sex halvsyskonfamiljer som förekom i provpopulationen var lätt urskiljbara grupper i analysen. Analysen för de andra sex tallarterna visade att SNP-chipet tappade i informativitet ju avlägsnare släkt arterna var med vår vanliga inhemska tall (Pinus sylvestris). Men även om Pinus-släktet är en stor grupp med 100 olika arter visade resultaten att 9 000 markörer var användbara, inte bara i Pinus-släktet utan även i Trifoliae och Strobus. För banksianatall, contortatall, svarttall och sydkinesisk tall var 10 841 SNPs av chipets totala 50 000 SNPs, fortfarande fullt användbara.
Resultat
Resultaten visar att ett SNP-chip på tall är en lovande markörresurs för både forskning och för skogsträdsförädling och att SNP-chipet har potential att ge ytterligare vinster till förädlingen, till exempel genom detaljerade släktskapsanalyser och genomiskt urval. Det krävs ytterligare forskning på och implementeringstester av markörer i genetisk utvärdering för att ännu bättre bestämma nyttan av DNA-markörer i förädlingen.
Lägesrapport för gran och björk
Utvecklingen av genomiska markörer pågår även för gran och björk inom förädlingsprogrammen. För gran finns ett SNP-chip med 50 000 markörer tillgängligt. Här fokuseras forskningen på att utöka antalet markörer för att förbättra precisionen i de genomiska avelsvärdena och möjliggöra genomisk selektion inom familjer.
För björk har vi inom projektet Trees for me utvecklat ett utvecklings-SNP-chip som har använts för att analysera över 400 individer. Nu pågår arbete med att välja ut de SNP som fungerar bäst, för att under 2025 ta fram ett slutgiltigt SNP-chip för genomisk selektion av björk.
Vi granskar och publicerar din kommentar så snart som möjligt.





